1991년 故 J. E. Bailey 박사가 대사공학의 개념을 정립한 이래 대사공학은 각종 실험적 기법의 결합과 함께 진화하여 왔고, 유전체,
전사체, 단백체, 대사체, 가상세포 시뮬레이션 등 다양한 오믹스 기법들의 발달은 기존 대사공학의 개념을 뛰어넘어 세포 전체의 관점에서 접근하는
시스템 대사공학으로 발전하였다. 대사공학으로 만들어진 각종 산업용 미생물 균주들은 식품, 바이오연료, 바이오플라스틱 등 이미 우리 실생활에 널리
이용되고 있다. 그러나 이러한 미생물 기반 화합물 생산 공정이 기존의 원유 기반 생산 공정을 대체하기 위해서는 보다 효율적인 대사경로 및 효소의
발굴 등을 통하여 균주의 생산성을 더욱 증가시켜야 한다 (Lee et al., 2012). 또한, 목표 화합물 또는 바이오매스의 화학적 가수분해
과정에서 발생하는 독성 물질에 대한 내성 등 단일 유전자 조작만으로는 개선하기 어려운 복합적인 형질들이 균주 개량에 필요한 시간을 더욱
소요시키는 주요한 원인으로 자리잡고 있다. 이러한 한계를 극복하기 위해서는 기존 자연계에서는 존재하지 않는 새로운 기작을 만들어 활용하거나,
자연계에 존재하는 기작을 우리가 원하는 방향으로 새롭게 재설계하는 합성생물학적인 기법의 도입 필요성이 대두되고 있다. 본 글에서는 최근
박테리아에서의 small regulatory RNA(sRNA)를 이용한 유전자 발현 조절 관련 연구의 동향과 이의 대사공학적 응용에 대하여
소개하고자 한다.
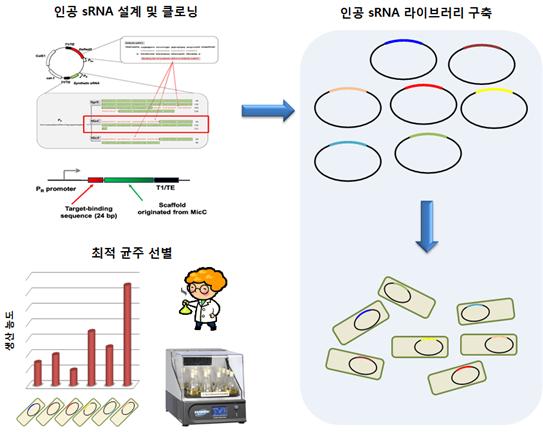
이러한 sRNA 기작을 공학적으로 이용한 사례로, 대장균의 Spot42 scaffold의 표적 결합 부위를 목표 유전자에 맞게 변이시킴으로써 ompF와 fliC의 발현 억제에 응용한 연구가 최근 소개된 바 있다(Shamar et al., 2012). 본 연구진에서도 이미 이러한 가능성에 주목하고, 기존의 Hfq-dependent sRNA scaffold를 목표 유전자에 맞게 재설계하여 성공적으로 목표 유전자 발현량 감소에 성공하였다. 이를 high-throughput 유전자 결실 목표 유전자 탐색에 응용하는 연구를 수행하여 유용 화학 물질인 L-타이로신과 나일론의 원료인 카다베린(cadaverine)의 고농도 생산 균주 제작에 성공하였으며, 그 결과의 중요성과 우수성을 인정받아 Nature Biotechnology (Na et al., 2013) 및 Nature Protocols (Yu et al., 2013)에 게재하였다 (그림 2).
우리가 원하는 물질을 고수율, 고농도로 생산하기 위해서는 불필요한 부산물의 생성에 관여하는 대사경로를 결실 또는 불활성화시키고, 목적
화합물 생합성 경로의 유전자를 증폭하는 것이 전형적인 방법일 것이다. 지금까지 미생물에서의 유전자 결실은 물리․화학적 돌연변이, 또는
전위요소(transposable element)를 도입하여 다양한 변이주를 만들고 여기에서 원하는 형질의 돌연변이주를 스크리닝하거나, 상동
재조합(homologous recombination) 등을 이용함으로써 목적 유전자만을 표적으로 하여 변이를 주는 방법 등이 주로 이용되어
왔다. 최근에는 게놈(genome)의 여러 목표 부위들을 변이시킬 수 있도록 single-stranded oligonucleotide
라이브러리를 구축하고, 이를 이용하여 우리가 원하는 형질을 갖는 변이주들을 제작하는 방법들이 보고된 바 있다(Wang et al., 2009;
Warner et al. 2010).
그러나 세포 전체의 대사 및 생리적 특성을 고려하지 못한 유전자 조작은 세포의 대사 균형을 무너뜨려 부작용을 야기할 수 있다. 대표적인
예로, 대장균 K12 균주의 아세트산 형성을 들 수 있다. 대장균에서 아세트산의 생성은 크게 피루브산 산화 효소(pyruvate
oxidase)를 거치는 경로와 아세틸 CoA로부터 인산:아세트산 전달 효소(phosphate acetyltransferase)와 아세트산
인산화 효소(acetate kinase)의 2단계 과정으로 생성되는 경로가 알려져 있다.이론적으로는 이 두 경로의 생합성 유전자 결실을 통하여
아세트산의 형성을 막을 수 있으며, 실제로도 그러한 보고가 있으나(Trinh et al., 2008), 탄소원, 목적 화합물, 발효 조건 및
모균주에 따라서 피루브산의 축적 또는 생장의 저해를 야기하기도 하며, 두 경로를 모두 불활성화시켜도 여전히 미량의 아세트산이 생성되는 결과가
발생하기도 한다(Causey et al. 2004). 이러한 경우에 대해서는 관련 유전자를 완전히 결실시키는 방법보다는, 생장기에 따라
선택적으로 아세트산 생합성 유전자 발현량을 조절하거나, 세포의 생장에 필요한 최소한의 양만 발현하게끔 조절하는 것이 더 바람직한 접근 방법이 될
수 있을 것이다.
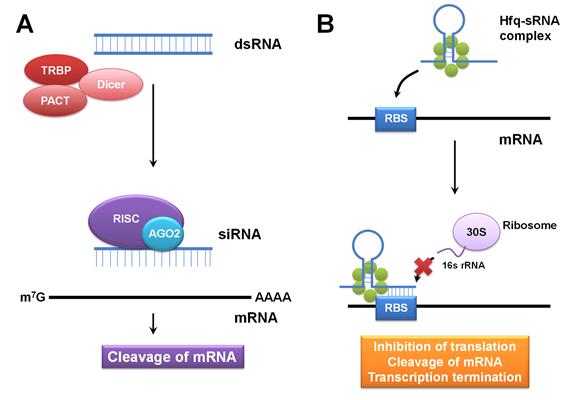
이러한 방법론 중의 하나가 유전자 침묵(gene silencing)이며, 진핵생물에서 RNA interference가 보고된 이래,
순수과학 및 공학 분야에서 small interfering RNA(siRNA; 그림 1A)와 microRNA 등의 non-coding RNA를
이용한 연구가 폭넓게 이루어지고 있으며, 부분적으로 그 기작도 규명되어있다.(Sablock et al., 2011). 따라서 본 글에서는
진핵생물에 비해 상대적으로 덜 알려진 원핵생물, 그 중에서도 박테리아의 small RNA(sRNA) 관련 연구에 관해 소개하고 이를 유전자 조작
도구로 활용한 사례에 대해 소개하고자 한다.
박테리아의 경우, 약 30년 전, 대장균에서 ompF의 발현량을 조절하는 micF RNA가 발견된 이후로
이미 antisense RNA(asRNA)를 유전공학적으로 사용하려던 시도가 많이 이루어졌으며, 이를 대사공학적인 도구로 활용한 예로는 유전자
조작이 어려운 것으로 알려진 부탄올 생산 미생물인 Clostridium acetobutylicum에서 사용한 경우가 대표적이다(Desai and
Papoutsakis, 1999). 근래에는 목표 유전자의 mRNA의 2차 구조를 분석하여 단일 가닥이 노출된 부분을 결합 부위로 삼아 in
vitro 실험으로 20 base 길이의 asRNA를 첨가하여 그 효과를 확인한 연구가 있으며(Shao et al., 2006), in
vivo에서 asRNA의 다양한 구조를 테스트하여 asRNA가 stem을 형성하면서 목표 mRNA와 결합하는 부위가 loop 형태로 노출될 때
발현 억제 효율이 가장 높았다는 연구가 보고된 바 있다(Nakashima et al., 2006; Nakashima et al., 2009).
그러나 실질적으로 이들 방법론을 통한 발현량의 감소 효율이 60-70%에 불과하다는 점에서 유전자 결실에 근접하는 발현량 감소로 보기에는
어려우며, 발현 감소량을 정밀하게 조절하는 도구로 활용하는 데에도 한계가 있다.

그림 1. (A) 진핵생물에서의 dsRNA에 의한 RNA interference의 모식도 (B) 대장균의 Hfq와
sRNA의 상호작용에 의한 유전자 발현 억제 과정의 모식도. sRNA와 Hfq 단백질이 결합하여 sRNA가 안정화되고 antisense 부위가
mRNA에 결합하여 번역 개시 억제, mRNA 절단, 또는 전사의 조기 억제를 유발한다.
최근의 연구 결과에 따르면 기존에 asRNA로 알려졌던 sRNA가 단독으로 유전자 발현을 저해하는 것이 아니라
단백질과의 상호작용을 통하여서도 이루어진다는 것이 밝혀졌다. 또한, 박테리아에서의 sRNA 수가 우리가 이제까지 생각했던 것보다 더욱 많이
존재하며, 유전자 발현을 증가시키거나 감소시킨다는 점에서 이들이 생리학적으로 중요한 역할을 한다는 것을 알 수 있다(Storz et al.,
2011). 대장균 및 이와 가까운 그람 음성균에서는 RNA chaperone으로 알려져 있는 Hfq가 sRNA와 상호작용을 함으로써 유전자
발현 조절이 일어난다고 알려져 있다 (그림 1B). 아직까지 Hfq와 sRNA가 유전자 발현 조절에 관여하는 기작은 정확하게 알려져 있지
않으나, 크게 (1) Hfq hexamer와 sRNA가 결합하여 sRNA를 RNase의 분해로부터 보호하거나 (2) Hfq hexamer 자체가
mRNA와 결합하여 mRNA의 분해를 촉진하는 기작이 존재하는 것으로 생각된다(Vogel and Luisi, 2011). 전자의 경우에 대해서는
sRNA의 서열에 따라 ribosome binding site(RBS)에 직접 결합하여 표적 mRNA의 번역 개시를 저해하거나,
double-stranded RNA-specific nuclease에 의한 분해 촉진, mRNA 전사의 조기 종결 등을 일으킴으로써 발현량을
감소시키는 것으로 추정하고 있다. 유전자 증폭의 경우에는 mRNA의 5’-UTR에 존재하는 anti-RBS 부위에 결합하여 RBS를
노출시킴으로써 번역을 촉진하는 경우가 알려져 있다(Vogel and Luisi, 2011).
이러한 sRNA 기작을 공학적으로 이용한 사례로, 대장균의 Spot42 scaffold의 표적 결합 부위를 목표 유전자에 맞게 변이시킴으로써 ompF와 fliC의 발현 억제에 응용한 연구가 최근 소개된 바 있다(Shamar et al., 2012). 본 연구진에서도 이미 이러한 가능성에 주목하고, 기존의 Hfq-dependent sRNA scaffold를 목표 유전자에 맞게 재설계하여 성공적으로 목표 유전자 발현량 감소에 성공하였다. 이를 high-throughput 유전자 결실 목표 유전자 탐색에 응용하는 연구를 수행하여 유용 화학 물질인 L-타이로신과 나일론의 원료인 카다베린(cadaverine)의 고농도 생산 균주 제작에 성공하였으며, 그 결과의 중요성과 우수성을 인정받아 Nature Biotechnology (Na et al., 2013) 및 Nature Protocols (Yu et al., 2013)에 게재하였다 (그림 2).

그림 2. 인공 sRNA 라이브러리를 이용한 대사공학 연구의 모식도
Bacillus, Corynebacterium, Streptomyces 등의 그람 양성균에서도 일부
sRNA가 밝혀져 학계에 보고되어 있으며, Clostridium 등의 비교적 원시적인 미생물에서도 sRNA가 존재한다는 것이
보고되었다는 점을 고려하면(Chen et al., 2011), sRNA에 의한 유전자 발현 조절은 거의 모든 박테리아에서 존재할 것으로
판단된다. 그러나 그람 양성균의 경우 대장균과 달리 목표 유전자 번역의 저해를 통한 조절이 아닌 mRNA의 2차 구조를 제거하는 것을 도와
번역을 촉진시켜 주는 형태의 기작을 가진 것이 상당수이며, Hfq homolog가 존재하지 않거나 있더라도 그 역할에 대해서는 아직 정확히
알려진 바가 없었다. 그러나 최근의 연구에서 그람 양성균인 Listeria monocytogenes에서도 Hfq의 영향을 받는
sRNA들이 존재하여 스트레스 내성과 병원성의 조절에 관여한다는 것이 밝혀졌으며 (Christiansen et al., 2006; Nielsen
et al., 2009), 최근 transcriptome 분석에 사용되는 RNA sequencing 등의 기술을 이용하면 게놈상의 small
RNA 코딩 부위 파악이 가능하므로, 그람 양성균의 sRNA 연구 또한 더욱 활발해질 것으로 기대한다. 다양한 종류의 sRNA들의 작용 기전이
규명된다면, 이를 우리가 원하는 표적 유전자에 특이적으로 작용하도록 새롭게 재설계함으로써 대사공학의 새로운 도구로 이용가능할 것이다. 특히,
산업 분야에서 많이 사용되는 미생물은 대장균에서 사용되는 다양한 유전자 조작 기술이나 high-throughput 기술을 적용하지 못하는 경우가
많으나, 인공 sRNA의 도입은 sRNA를 발현할 벡터만 도입하면 충분히 가능하므로 산업용 균주 개발에 있어서도 그 응용 가치가 무궁무진할
것이다. 또한 이러한 도구를 합성 유전자 회로와 연계함으로써 세포의 외부 환경에 따라 특정 유전자들의 발현량 증폭 또는 감소를 유도함으로써
대사의 강건성(robustness)이 향상된 균주의 개발에도 활용할 수 있을 것이다.
참고문헌
-Ajikumar, P. K., Xiao, W. H,, Tyo, K. E., Wang, Y., Simeon, F.,
Leonard, E., Mucha, O., Phon, T. H., Pfeifer, B., and Stephanopoulos, G. (2010)
Isoprenoid pathway optimization for Taxol precursor overproduction in
Escherichia coli. Science 330(6000):70-4.
-Causey, T. B., Shanmugam, K. T., Yomano, L. P., and Ingram L. O.
(2004) Engineering Escherichia coli for efficient conversion of glucose to
pyruvate Proc. Natl. Acad. Sci. USA. 101(8): 2235-40.
-Chen, Y., Indurthi, D. C., Jones, S. W., and Papoutsakis, E. T.
(2011) Small RNAs in the genus Clostridium. MBio. 2011 2(1):e00340-10.
-Choi, J. H., Keum, K. C., and Lee, S. Y., Production of
recombinant proteins by high cell density culture of Escherichia coli. (2006)
Chem. Eng. Sci., 66: 876-85.
-Christiansen, J. K, Nielsen, J. S, Ebersbach, T.,
Valentin-Hansen, P., Søgaard-Andersen, L., Kallipolitis, B. H., Identification
of small Hfq-binding RNAs in Listeria monocytogenes. (2006) RNA. 12(7): 1383-96.
-Desai, R. P., and Papoutsakis, E. T. (1999) Antisense RNA
strategies for metabolic engineering of Clostridium acetobutylicum. Appl Environ
Microbiol. 65(3):936-45.
-Lee, J. W., Na, D., Park, J. M., Lee, J., Choi, S., and Lee, S.
Y. Systems metabolic engineering of microorganisms for natural and non-natural
chemicals (2012) Nat. Chem. Biol. 8(6): 536-46.
-Na, D., Yoo, S. M., Chung, H., Park, H., Park, J. H., Lee S. Y.,
Metabolic engineering of Escherichia coli using synthetic small regulatory RNAs.
(2013) Nat. Biotechnol. 31(2):170-4.
-Yoo, S. M., Na, D., Lee, S. Y. Design and use of synthetic
regulatory small RNAs to control gene expression in Escherichia coli. (2013) Nat
Protoc. 8(9):1694-707.
-Nakashima, N., Tamura, T., and Good, L. (2006) Paired termini
stabilize antisense RNAs and enhance conditional gene silencing in Escherichia
coli. Nucleic Acids Res. 34(20):e138.
-Nielsen, J. S., Lei, L. K., Ebersbach, T., Olsen, A. S.,
Klitgaard, J. K., Valentin-Hansen, P., Kallipolitis, B. H. Defining a role for
Hfq in Gram-positive bacteria: evidence for Hfq-dependent antisense regulation
in Listeria monocytogenes. (2010) Nucleic Acids Res. 38(3): 907-19.
-Sablok, G., Pérez-Quintero, A. L., Hassan, M., Tatarinova, T. V.,
and López, C. (2011) Artificial microRNAs (amiRNAs) engineering - On how
microRNA-based silencing methods have affected current plant silencing research.
Biochem. Biophys. Res. Commun. 406(3):315-9.
-Shao, Y., Wu, Y., Chan, C. Y., McDonough, K., and Ding, Y. (2006)
Rational design and rapid screening of antisense oligonucleotides for
prokaryotic gene modulation. Nucleic Acids Res. 34(19):5660-9.
-Sharma, V., Yamamura, A., and Yokobayashi, Y. (2012) Engineering
Artificial Small RNAs for Conditional Gene Silencing in Escherichia coli. ACS
Synth. Biol. 1(1):6–13.
-Storz, G., Vogel, J., Wassarman, K. M. (2011) Regulation by small
RNAs in bacteria: expanding frontiers. Mol. Cell 43(6):880-91.
-Trinh, C. T., Unrean, P., and Srienc F. (2008) Minimal
Escherichia coli cell for the most efficient production of ethanol from hexoses
and pentoses. Appl. Environ. Microbiol. 74(12):3634-43.
-Vogel, J., Luisi, B. F. (2011) Hfq and its constellation of RNA.
Nat. Rev. Microbiol. 9(8):578-89.
-Wang, H. H., Isaacs, F. J., Carr, P. A., Sun, Z. Z., Xu, G.,
Forest, C. R., and Church, G. M. (2009) Programming cells by multiplex genome
engineering and accelerated evolution. Nature 460(7257):894-8.
-Warner, J. R., Reeder, P. J., Karimpour-Fard A., Woodruff, L. B.,
Gill, R. T. (2010) Rapid profiling of a microbial genome using mixtures of
barcoded oligonucleotides. Nat. Biotechnol. 28(8):856-62.
출처 : 한국과학기술원 이상엽












