대장균
ncRNA의 합성 생물학 및 이의 응용
이영훈 교수
한국과학기술원
1. 개요
DNA가 담고 있는 유전 정보를 단백질로 전달하는 기능을 하는 생체분자인 RNA가 그 자체로써 여러 기능을 수행하고 있음이 밝혀지고
있다. 단백질을 코딩하지 않는 RNA (non-coding RNA: ncRNA)라 불리는 이 RNA는 다양한 생명 현상에 능동적으로 관여하고
있고 RNA 간섭현상 (RNA interference), miRNA에 의한 유전자 발현 조절과 이를 응용한 많은 연구방법이 개발되면서
ncRNA에 대한 연구는 크게 주목 받고 있다. 박테리아의 40~500 nt 길이를 가지는 100종 이상의 다양한 ncRNA가 존재하고 있고,
많은 연구가 대장균을 모델로 하여 ncRNA가 세포내에서 어떤 메커니즘으로 어떤 기능을 하는지 하는 연구에 초점을 맞추어 수행되고 있다.
최근에는 ncRNA의 기능중의 하나인 유전자 침묵 (gene silencing)을 이용하여 유전자 발현을 조절하고 이를 세포대사공학에 응용하는
연구가 시도되고 있다. 그러나 효과적이고 보다 정교한 방법으로 ncRNA를 세포대사공학에 활용하기 위하여 ncRNA에 의한 유전자 침묵의
메커니즘의 규명이 필요하다. 본 연구 동향에서는 유전자 침묵을 위한 효과적인 인공 ncRNA의 디자인 방법과 ncRNA 기능에 영향을 미치는
RNA 나선효소 (RNA helicase)에 대한 연구를 소개하려 한다.
2. 인공 ncRNA의 디자인 및 타겟 인식
연구
대장균에서 ncRNA는 대장균이 다양한 성장환경변화에 대응하는데 중심 조절인자로 작용하고 있다. ncRNA는 전에 예상하였던 것 보다 종류가 많고, 세포내 다양한 대사작용 조절 네트워크에서 중요한 역할을 한다는 것이 밝혀지고 있다. 그러나 세포내에서 ncRNA가 작용하는 정확한 메커니즘은 분석 방법의 어려움으로 완전히 이해되지 않고 있다. 특히 ncRNA에 의한 유전자 침묵은 RNA-RNA 상호작용으로 일어나는데, ncRNA와 mRNA 사이의 RNA-RNA 상호작용을 통한 유전자 침묵의 상세한 메커니즘은 여전히 불분명하다. 최근 연구에서 인공 ncRNA인 afsRNA (artificial sRNA)를 이용하여 세포내에서 유전자 침묵에 필요한 염기쌍 형성의 메커니즘이 조사되었다. afsRNA는 대장균 sRNA인 SibC RNA의 골격을 이용하였고, 여기에 다양한 타겟인식 염기순서를 도입하였다 (그림 1). 이 연구에서 개발된 afsRNA의 특징은 다른 인공 sRNA와 달리 다양한 타겟인식 염기순서를 도입하여도 전체 afsRNA 구조에 큰 변화가 없고, 타겟인식 염기순서는 afsRNA의 단일가닥 영역에 존재한다는 것이다. afsRNA를 이용하여 먼저, 효과적인 유전자 침묵에 필요한 최소 염기쌍 수는 10개라는 것을 밝혔다. 또한, sRNA와 mRNA 사이에 잘못 짝지은 염기쌍이 존재하면 염기쌍의 수가 같아도 afsRNA에 의한 유전자 침묵은 감소된다는 밝혔고, 잘못 짝지은 경우에도 내부 고리 (internal loop)를 형성하는 afsRNA가 돌출부 (bulge)를 형성하는 afsRNA 보다 유전자 침묵을 수행하는데 더 효과적이라는 것을 밝혔다. 특히, 본 연구에서 개발한 afsRNA는 hfq 돌연변이에 의하여 유전자 침묵이 감소되지 않았고, 오히려 afsRNA와 mRNA 사이의 염기쌍 형성이 약할 경우 증가하였다. 이 결과는 염기쌍 형성이 약할 때 Hfq가 유전자 침묵을 억제한다는 것을 의미 하는 것으로 지금까지 알려진 Hfq의 기능, 즉 유전자 침묵을 증가시키는 기능과 반대되는 결과이다. 염기쌍이 약할 때 유전자 침묵을 억제하는 Hfq의 기능은 짧은 염기쌍 형성으로 원하지 않는 타겟인식에 의하여 야기될 수 있는 ncRNA의 과녁을 벗어난 효과 (off-target effect)를 감소시키는 역할을 할 지 모른다. 그러므로 이러한 연구 결과는 세포내에서 RNA-RNA 상호작용을 통하여 일어나는 ncRNA의 작용메커니즘을 좀 더 깊이 이해하고, ncRNA를 정교하게 디자인하여 세포대사공학에 효과적으로 활용하는데 기여할 수 있을 것으로 예상된다.
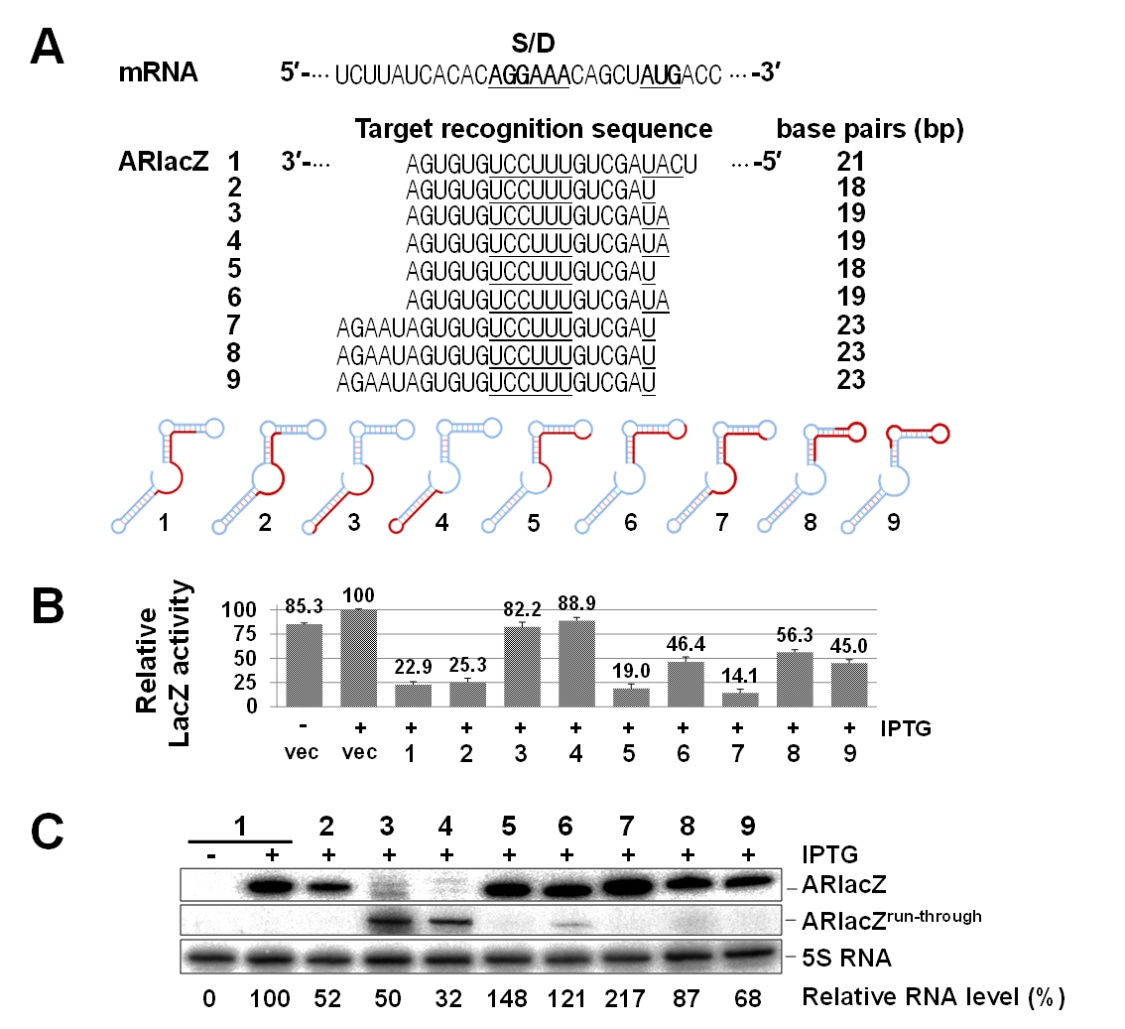
그림 1. 인공 ncRNA인 afsRNA의 다양한 구조와 이들에 의한 유전자 침묵 효과. (A) lacZ mRNA를
인식하는 afsRNA인 ARlacZ
의 타겟 인식 염기서열. RNA 이차구조에서 타겟 염기서열은 빨간색으로 표시. (B) 유전자 침묵효과. (C) 세포내에서
인공 ncRNA 발현 확인. Hongmarn Park, et al. Nucleic Acids Res. 41(2013):
3787-804에서.
3. ncRNA 작용에서 RNA 나선효소(helicase)의
역할
RNA가 세포내에서 작용을 할 때 RNA 구조는 매우 중요하다. RNA 이차 구조는 이중가닥과 단일가닥 영역으로 나타낼 수 있는데 이중가닥은 RNA 나선효소에 의하여 단일가닥으로 변할 수 있다. ncRNA의 타겟이 RNA인 경우 ncRNA는 타겟 RNA와 이중나선 (helix)을 형성할 수 있다. 이 이중 나선의 형성 속도는 ncRNA 기능에 중요한 역할을 한다. RNA 나선효소는 ncRNA의 이차구조는 물론 타겟 RNA의 이차구조를 변화시킴으로써 ncRNA에 의한 타겟 RNA 인식 속도를 변화시킬 수 있다. 또한 형성된 ncRNA-타겟 RNA 복합체도 RNA 나선효소에 의하여 다시 분해될 수 있다. 그러므로 RNA 나선효소가 ncRNA 기능에 많은 영향을 미칠 것으로 예상할 수 있다. 대장균에는 5종의 RNA 나선효소가 존재한다. DbpA, DeaD (CsdA), SrmB, RhlB, RhlE 5종 중 DeaD, SrmB, RhlE는 저온 충격 (cold shock) 단백질로도 알려져 있다. 성장정지기 (stationary phase)에서 RNA 중합효소의 시그마 성분 (sigma subunit)인 σS (rpoS 유전자 산물)의 발현은 ncRNA인 DsrA에 의하여 활성화 되는데 DeaD RNA 나선효소가 과발현될 때 DsrA에 의한 rpoS의 발현이 더 활성이 된다 (2). DsrA는 rpoS mRNA의 변역개시영역 (translation initiation region)에 결합하여 rpoS mRNA의 이차구조를 변형시켜 rpoS mRNA의 S/D 염기서열과 변역개시 코돈을 리보솜에 노출시킴으로써 rpoS 발현을 활성화 시킨다. 그러므로 DeaD에 의한 DsrA-매개 rpoS 활성화가 증가되었다는 결과는 DeaD RNA 나선효소가 DsrA의 2차 구조를 풀어줌으로써 rpoS mRNA과 DsrA의 상호작용이 촉진되었다는 것을 시사한다 (그림 2A).
오래 전부터 ncRNA로 잘 알려진 rRNA 기능도 RNA 나선효소에 의하여 영향을 받는다. rRNA는 일차전사체로 전사된 선구 rRNA (pre-rRNA)가 여러 리보핵산 가수분해효소에 의해 5’, 3’ 말단이 가공되어 성숙된 rRNA로 만들어 진 후 리보솜 단백질들과 결합하여 리보솜 소단위체 (subunit)를 형성한다. 이 때 rRNA가 정확히 가공되지 못하면 리보솜 조립 (ribosome assembly)에 결함이 생기게 되는데, rRNA 가공가정에 RNA 나선효소가 관련되어 있다는 연구 결과가 최근에 발표되었다 (3). 이는 DeaD RNA 나선효소가 없을 때 50S 리보솜 단위체를 이루는 23S rRNA가 제대로 가공이 되지 않기 때문이다. 또한 16S rRNA의 경우에는 3’ 말단 가공과정에 참여하는 리보핵산 가수분해 효소인 RNase R과 RMF (ribosome maturation factors) 이외에도 YbeY 단백질이 리보핵산 가수분해효소로서 손상된 16S rRNA를 제거하여 리보솜 품질관리를 하는 기능을 한다는 것이 밝혀졌는데 (4) 이때에도 RNA 나선효소가 중요한 역할을 한다. 그러므로 유전자 발현조절 및 세포대사공학에 ncRNA를 활용하려는 연구를 수행할 때, RNA 나선효소의 역할을 고려할 필요가 있을 것으로 보인다.
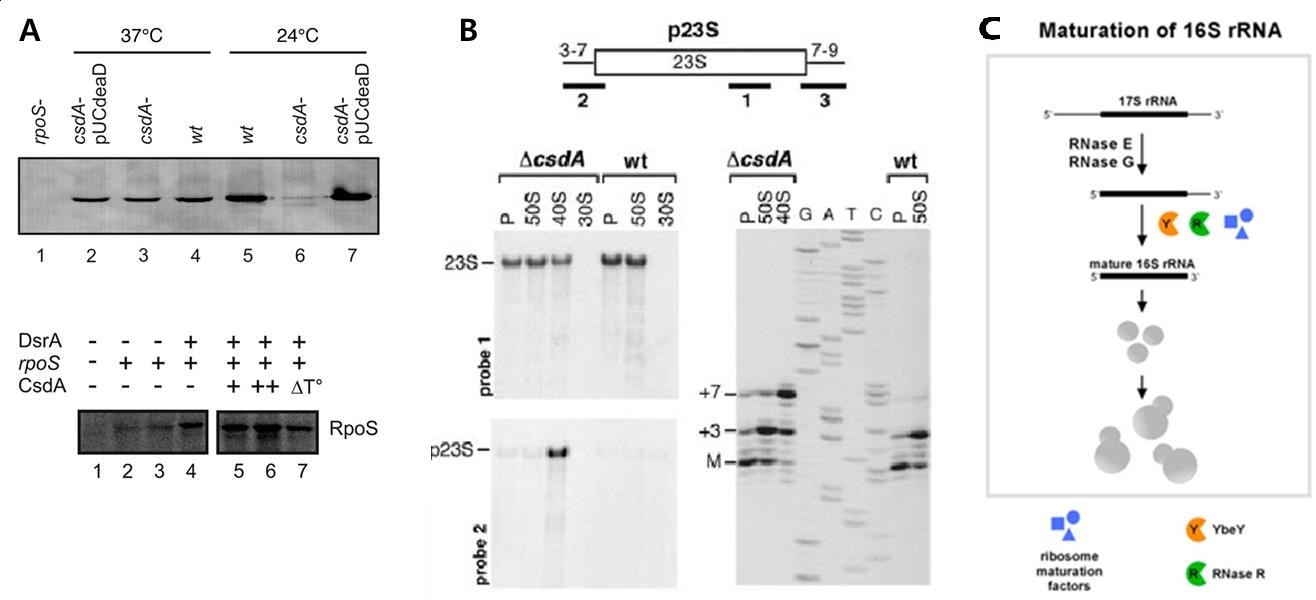
그림 2. (A) RpoS 생합성에 관여하는 DsrA ncRNA의 저온에서의 기능 강화에 필요한 CsdA (DeaD) [Armin Resch et al. RNA
Biology 7(2010): 1-7에서]. (B) 23S rRNA의 성숙과정에서 CsdA의 역할 [Julie
Charollais et al. Nucleic Acids Res. 32
(2004): 2751-2759에서]. (C) 16S rRNA 성숙모델에서 RMF (ribosome maturation
factors)로서 RNA 나선효소의 역할
[Asha Ivy Jacob et al. Molecular Cell. 49(2013):
427–438에서].
References
1. Hongmarn Park et al. 2013. Exploring sRNA-mediated gene silencing
mechanisms using artificial small RNAs derived
from a natural RNA scaffold in Escherichia coli. Nucleic Acids Res. 41:
3787-3804.
2. Armin Resch et al. 2010. Requirement of the CsdA DEAD-box helicase for low temperature riboregulation of rpoS mRNA.
2. Armin Resch et al. 2010. Requirement of the CsdA DEAD-box helicase for low temperature riboregulation of rpoS mRNA.
RNA Biology 7: 1-7.
3. Julie Charollais et al. 2004. CsdA, a cold-shock RNA helicase from Escherichia coli, is involved in the biogenesis of
3. Julie Charollais et al. 2004. CsdA, a cold-shock RNA helicase from Escherichia coli, is involved in the biogenesis of
50S ribosomal subunit. Nucleic Acids Res. 32: 2751-2759.
4. Asha Ivy Jacob et al. 2013. Conserved bacterial RNase YbeY plays key roles in 70S ribosome quality control and 16S
4. Asha Ivy Jacob et al. 2013. Conserved bacterial RNase YbeY plays key roles in 70S ribosome quality control and 16S
rRNA maturation. Molecular Cell. 49: 427–438.












